
发布日期:2025-12-03浏览次数:216来源:蓝景科信
2025年6月12日,美国麻省理工和博德研究所Pardis C.Sabeti团队在Science上发表了一篇题为“Pan-viral ORFs discovery using massively parallel ribosome profiling”的论文,结合寡核苷酸合成文库和Ribo-seq核糖体印迹分析,开展泛病毒ORF挖掘,本研究旨在建立一种高效、无偏的泛病毒ORFs发现方法,填补病毒翻译组学空白。

尽管病毒基因组测序技术已有很大进展, 但病毒基因组的功能注释长期滞后于测序进展。除了已注释的经典开放阅读框(ORFs)之外,尤其是不符合传统ORF的定义的非经典ORFs(如非ATG起始、长度<100aa)在感染、免疫调控中的作用尚未系统探索。
传统的病毒基因组注释方法主要依赖于生物信息学预测,存在假阳性率高、无法区分功能性和非功能性ORFs等局限性。更重要的是,许多高致病性病毒(如埃博拉病毒、尼帕病毒等)难以在实验室条件下培养,需要在生物安全三级(BSL-3)或四级(BSL-4)设施中操作,极大限制了对这些病毒基因组的深入研究。
核糖体印迹分析也称为Ribo-seq,此项技术的发展极大提升了检测基因组翻译区域的能力。该技术利用对核糖体结合的RNA片段进行深度测序,确定核糖体占位情况,从而以单核苷酸分辨率揭示翻译区域。它已经在哺乳动物细胞、酵母、细菌和病毒中发现了许多非经典ORFs,包括5′非翻译区(5′UTRs)中的上游ORFs(uORFs)和上游重叠ORFs(uoORFs)、非编码RNA中的短ORFs以及已注释编码序列中的重叠内部ORFs(iORFs)。
然而,传统核糖体图谱技术在病毒研究中面临三大瓶颈:病毒培养困难、安全要求高、遗传多样性强。每种病毒需要特定的培养系统,部分病毒无法在实验室培养;高致病性病毒需要高等级生物安全设施;单一病毒株分析无法覆盖变异谱系,导致全球多数病毒的翻译图谱仍是"黑箱"。
为解决这些挑战,博德研究所的Shira Weingarten-Gabbay和Pardis C. Sabeti团队开发了大规模平行核糖体图谱(MPRP)技术。该技术创新性地将高通量寡核苷酸合成与核糖体图谱技术相结合,在一次实验中可同时分析数百种病毒的翻译事件,无需病毒培养或高生物安全等级设施,极大降低了实验门槛。这一技术突破为病毒基因组研究提供了全新的方法学工具,有望彻底改变我们对病毒蛋白质组的认知。
MPRP技术的核心原理是利用合成寡核苷酸文库替代活病毒,通过Ribo检测病毒基因组片段的翻译活性。
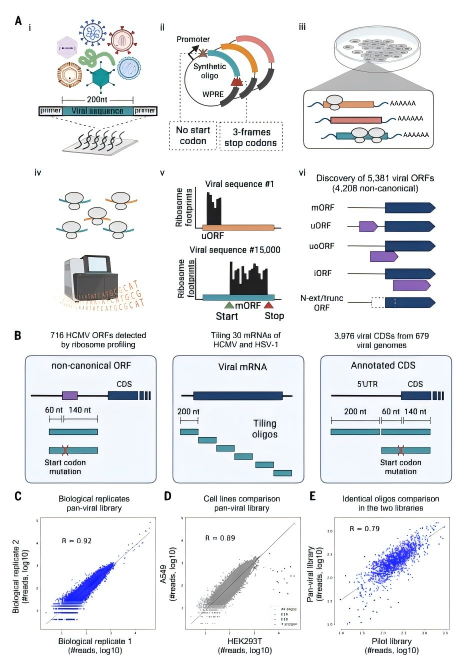
1.文库构建:研究团队设计了包含20,170个合成寡核苷酸的文库,覆盖679种人类相关病毒基因组的3,976个基因的5'非翻译区(5'UTR)和编码区起始区域。每个寡核苷酸包含60个核苷酸的5'UTR和140个核苷酸的编码区,确保能够捕获翻译起始位点和上游调控序列。
2.并行检测:将文库转染至HEK293T/A549细胞,在病毒感染相关应激条件(如poly(I:C)、内质网应激)下进行核糖体分析,使用抑制剂区分起始/延伸核糖体。
3.数据处理:采用PRICE(Probabilistic Ribo-seq Inference of Coding Exons)算法从核糖体足迹数据中推断ORFs,并通过免疫肽组学验证HLA-I呈递。
4.跨平台验证:对比MPRP数据与HCMV、VSV等天然感染细胞的Ribo-seq结果,确保可靠性。
3.1 病毒ORFs的大规模发现
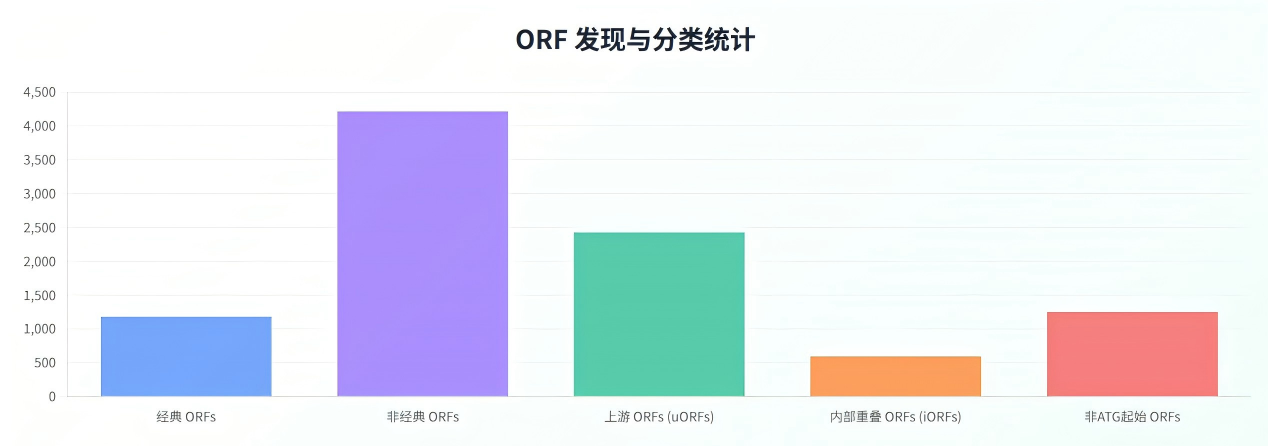
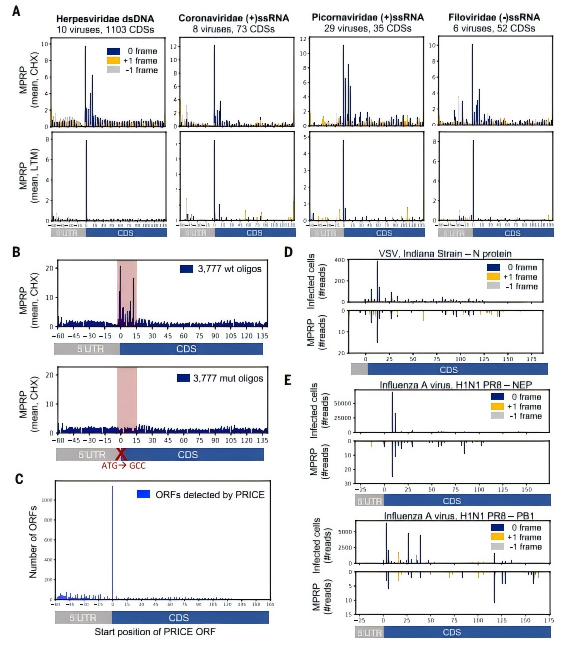
MPRP技术在679种病毒中实现了数量级的发现突破,共鉴定出5,381个病毒ORFs,其中4,208个为非经典ORFs,覆盖疱疹病毒、冠状病毒、丝状病毒等21个病毒家族。这些新发现的ORFs极大扩展了我们对病毒蛋白质组的认知边界,揭示了病毒基因组中存在大量此前未被注释的翻译区域。
ORF类型的多样性令人瞩目。研究发现了多种类型的非经典ORFs:上游ORFs(uORFs)位于5'UTR区域,如HCMV UL45 5'UTR的uORF可导致核糖体停滞,抑制主CDS翻译;内部重叠ORFs(iORFs)如IAV M1基因+1框架的iORF,在H5N1等6种流感毒株中保守存在;非ATG起始ORFs如HCMV中以GUG、UUG起始的ORFs,编码20个氨基酸以下的微蛋白。这些不同类型的ORFs可能在病毒生命周期中发挥多样化的功能。
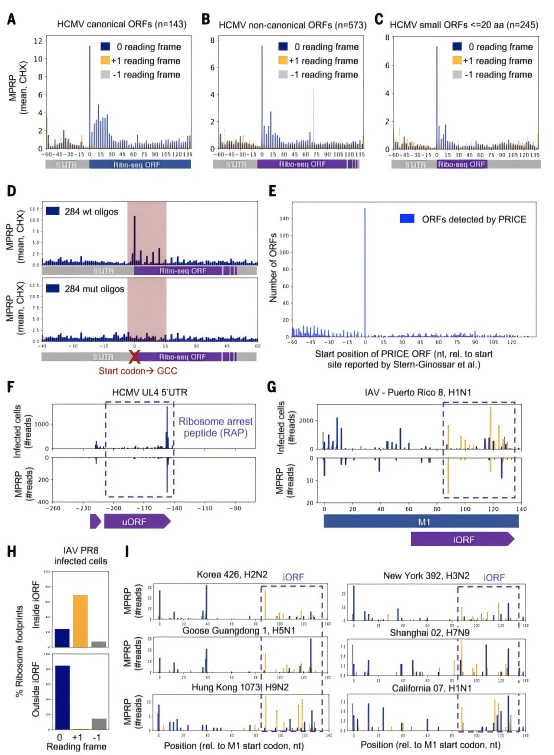
技术性能评估显示,在3,976个注释的病毒CDS中,PRICE算法成功捕获了1,136个(28%)在注释起始密码子处起始的ORFs,错误发现率为0.05。虽然捕获率看似不高,但考虑到实验系统的复杂性和病毒基因组的多样性,这一结果仍具有重要意义。更重要的是,PRICE检测到的CDS在两个生物学重复中的核糖体占用率显示出高相关性(R=0.93),证明了检测结果的可靠性。
3.2 非经典ORFs的免疫原性验证
研究的一个重要发现是非经典ORFs具有显著的免疫原性。通过分析感染细胞的免疫肽组数据集,研究团队发现MPRP鉴定的非经典ORFs能够产生人类白细胞抗原I类(HLA-I)相关肽段。在HCMV中发现了4个非经典ORF来源的5种肽段,如UL4 5'UTR uORF的VLSAKKLS、UL135 5'UTR uoORF的YPAPRPQAI;在VACV中发现了2种肽段,如L3L iORF的HRNKIINAEK,这些肽段均被HLAthena预测为高亲和力HLA-I结合者(MSi rank ≤2)。
免疫贡献的量化分析揭示了非经典ORFs的重要性。将MPRP鉴定的ORF纳入HCMV注释后,HLA-I呈递肽段数量增加7.4%,且非经典ORF的单位长度产肽量显著高于经典ORF(P<10⁻³)。这一发现表明,非经典ORFs不仅能够编码功能性肽段,还具有更高的免疫原性密度,可能在病毒免疫逃逸和宿主免疫应答中发挥重要作用。
3.3 uORF对病毒翻译的调控机制
研究揭示了上游ORFs(uORFs)对病毒蛋白翻译的精细调控机制。在正常条件下,2,418个病毒基因中37%的核糖体聚集在5'UTR(uORF区域),抑制主CDS翻译;亚砷酸钠诱导eIF2α磷酸化后,核糖体向CDS转移,5'UTR聚集比例降至19%,证实uORF通过eIF2α通路调控翻译起始。
功能保守性证据表明uORF的调控作用在不同实验条件下具有一致性。HCMV UL4 5'UTR的uORF在MPRP和天然感染细胞中均导致核糖体停滞,其编码的核糖体停滞肽(RAP)可抑制主蛋白翻译。三核苷酸周期性分析显示,在5'UTR区域检测到的uORFs具有活跃的核糖体翻译特征,且在LTM抑制剂存在下,这些uORFs的起始密码子处显示强烈的起始核糖体信号。
研究还发现,在应激条件下(如eIF2α磷酸化),核糖体更容易绕过5'UTR中的uORFs,在主CDS处起始翻译,这与抑制性uORF的预期行为一致。这一发现为理解病毒如何在不同细胞环境中调控蛋白合成提供了重要见解,也为开发针对病毒翻译调控的治疗策略提供了潜在靶点。
MPRP技术为病毒基因组研究带来了范式转变,通过揭示病毒基因组中大量"隐藏"的ORFs, 不仅扩展了病毒蛋白质组的边界,更为疫苗开发、抗病毒药物研发和疫情防控提供了重要机遇。 这一技术突破不仅是科学技术的进步,更是人类抗击病毒的重要武器。
